学术进展丨我院秦国友、余勇夫团队在高维数据迁移学习方法研究领域取得进展
近日,复旦大学公共卫生学院秦国友、余勇夫团队联合山西医科大学公共卫生学院王彤教授团队在生物信息学权威期刊《PLOS Computational Biology》上在线发表了题为“A robust transfer learning approach for high-dimensional linear regression to support integration of multi-source gene expression data”的研究论文。该研究提出了提出了一种具有t分布误差的高维线性模型的迁移学习框架,为提升重尾分布和异常值条件下的数据估计与预测精度提供了新的解决方案。

随着基因组技术的快速发展,公共数据库中积累了大量可用于生物医学研究的基因表达数据。多源数据的整合应用不仅能够有效弥补目标数据样本量不足的局限,更为深入探索基因调控机制、揭示疾病发生发展规律提供了重要机会。然而,不同源数据间的异质性使得有效利用源数据中的信息存在困难。迁移学习方法的引入为解决这一难题提供了可能。
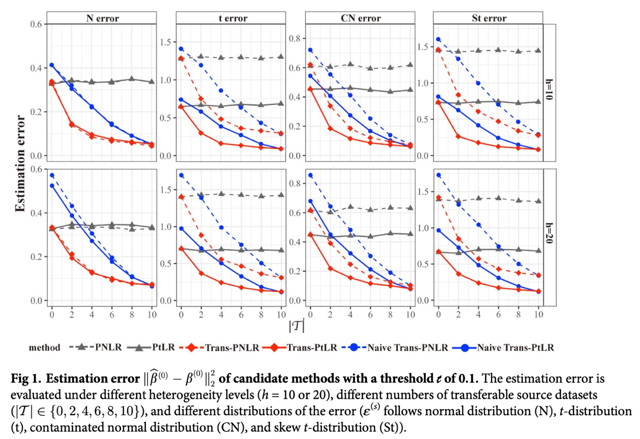
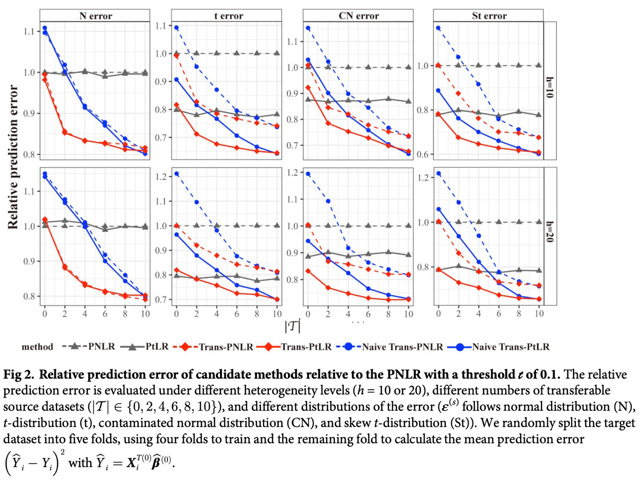
然而,基因组数据中普遍存在的重尾分布和异常值现象对现有迁移学习方法的有效性提出了挑战。为此,团队创新性地构建了具有t分布误差的高维线性模型(Trans-PtLR),通过借用源数据中的有用信息,显著提升了模型对重尾分布和异常值数据的鲁棒性。研究采用惩罚最大似然与期望最大化算法相结合的策略,构建了迁移学习算法框架,并引入基于交叉验证的源数据筛选机制,有效避免了不可迁移的源数据的干扰。
通过大量模拟实验和实际应用验证,研究证实Trans-PtLR模型相较于传统基于正态误差分布的线性回归迁移学习方法具有显著优势。特别是在GTEx数据库的应用研究中,团队以JAM2基因为研究对象,选取1292个相关基因作为预测因子,将13种脑组织作为目标组织,其余36种组织作为源组织进行建模分析。实验结果表明,该研究方法在所有目标组织中的预测准确率均优于现有方法,展现出显著的性能优势。这一研究成果为基因组数据的整合分析提供了新的方法学工具。
复旦大学公共卫生学院二年级硕士生潘璐璐、山西医科大学公共卫生学院高倩副教授、复旦大学公共卫生学院博士生魏可成为本文共同第一作者,复旦大学公共卫生学院余勇夫教授、复旦大学公共卫生学院秦国友教授、山西医科大学公共卫生学院王彤教授为共同通讯作者。该研究得到了国家自然科学基金(82173612;82273730;82073674;82373692;82204163)、上海市青年科技启明星计划(21QA1401300)、上海市自然科学基金(22ZR1414900)、上海市市级科技重大专项(ZD2021CY001)和山西省基础研究计划(202203021212382)的资助。
论文链接:https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1012739








